Identify histone modification profiles on a genome-wide scale.
Genome-scale analysis of transcription factor binding sites.
Advanced ChIP-Seq normalization strategy helps reveal hidden global changes.
Bioinformatics approach to identify master regulators of gene expression.
Obtain high-quality ChIP-Seq data from valuable clinical FFPE samples.
客户对我们的评价
"Active Motif花时间了解我们的问题,了解了我们的目标并讨论了所有选项和含义。 不仅仅通过ChIP-Seq实验验证了我们的数据,而且还强调了一种新的机理。 数据和服务的质量惊人地好。 优秀的科学知识,知识渊博且乐于助人的人员和优质的数据。"
David Briere, PhD
首席科学家
Mirati Therapeutics
查看完整的客户评价 >
ChIP-Seq服务包括:
客户提供固定的细胞沉淀或冷冻组织,然后我们:
- 准备染色质样品并超声处理。
- 使用验证过的抗体进行ChIP实验。
- 建立ChIP-Seq测序文库。
- 进行二代测序。
- 分析并发送数据。
ChIP-Seq Services Sample Submission Portal
Our online sample submission portal allows you to easily upload your service project samples and track your project status. Follow the sample submission instructions in the portal to ensure that all your samples arrive at Active Motif in the best possible condition and properly associated with your project.
要了解更多信息,请电话联系我们或向我们发送表观遗传服务信息申请。 您还可以下载Active Motif的表观遗传服务介绍。
| Name | Cat No. | Price | |
|---|---|---|---|
| FactorPath™ ChIP-Seq | 25200 | Get Quote | |
| HistonePath™ ChIP-Seq | 25215 | Get Quote | |
| TranscriptionPath™ ChIP-Seq | 25220 | Get Quote |
在ChIP-Seq中,得到数据只是成功的一半。 测序完成后,必须将数千万个短序列标签比对到基因组,然后进行peak calling 。peak calling非常复杂,根据所使用的抗体,需要不同的算法来进行准确的peak calling。 然后必须将成千上万的结合位点输出到有意义的结果中,将数据与基因相关联,并允许多个样本相互比较。 聚类,热图和全基因组定位模式的可视化呈现仍面临挑战。 这种类型的深度生物信息学分析超出了大多数实验室的能力。 因此,数据分析是我们所有ChIP-Seq服务项目的标准服务的一部分。
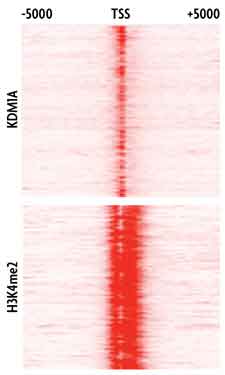
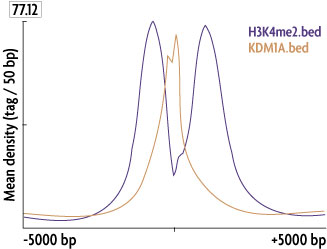
图3:相对于基因转录起始位点(TSS)的所有基因组结合位点的汇集。
用组蛋白脱甲基酶KDM1A及其靶标之一的组蛋白H3K4me2抗体进行ChIP-Seq实验。 因为与基因注释有关,鉴定了数千个结合位点,并汇集了所有结合位点的位置。左图。使用由H3K4me2和KDM1A结合的所有基因创建了一个热图,并描述了转录起始位点(TSS)周围这些因子的结合率。上图。相同的数据使用不同方式显示KDM1A 在H3K4me2结合的两个峰之间的结合率。
以下论文引用了Active Motif表观遗传服务提供的有关ChIP-Seq服务和/或其他信息:
- “Reduced H3K27me3 and DNA Hypomethylation Are Major Drivers of Gene Expression in K27M Mutant Pediatric High-Grade Gliomas” by Bender et al (2013) Cancer Cell 24(5):660-672.
- “Atrial Identity Is Determined by a COUP-TFII Regulatory Network” by Wu et al (2013) Developmental Cell 25(4):417-426.
- “Specification of type 2 innate lymphocytes by the transcriptional determinant Gfi1” by Spooner et al (2013) Nature Immunology 14(2):1229-1236.
- “EZH2 inhibition as a therapeutic strategy for lymphoma with EZH2-activating mutations” by McCabe et al (2012) Nature 492(7427):108-112.
- “Loss of the Tumor Suppressor BAP1 Causes Myeloid Transformation” by Dey et al (2012) Science 337(6101):1541-1546.
- “ChIP sequencing of cyclin D1 reveals a transcriptional role in chromosomal instability in mice” by Casimiro et al (2011) Journal of Clinical Investigation 122(3):833-843.
- “Global Characterization of Transcriptional Impact of the SRC-3 Coregulator” by Lanz et al (2010) Molecular Endocrinology 24(4):859-872.
- “A ChIP-seq defined genome-wide map of vitamin D receptor binding: Associations with disease and evolution” by Ramagopalan et al (2010) Genome Research 20(10):1352-1360.
- “miR-155 Inhibition Sensitizes CD4+ Th Cells for TREG Mediated Suppression” by Stahl et al (2009) PLoS ONE 4(9):e7158.

